- 文/張家銘│ 政治大學資訊科學系生物資訊實驗室,希望透過資訊方法的幫忙,來一探生物之秘。
收納大作戰,兩公尺長的遺傳資訊怎麼收?
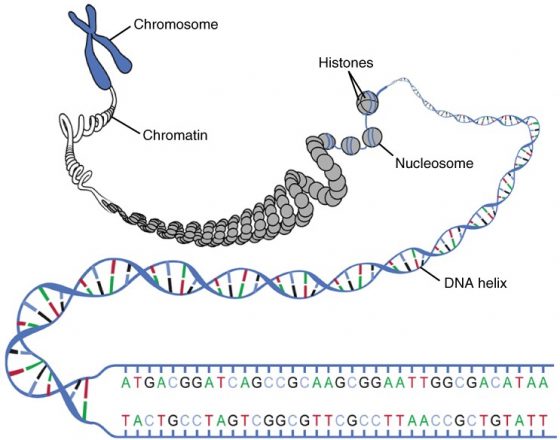
我們知道每個細胞攜帶 23 對染色體,而這遺傳資訊儲存在細胞核中,相較於 6 微米的細胞核,把存在其中的 DNA 資訊拉直約有 2 公尺長,這有多麼不可思議? 有如把繞地球 3 百多萬圈的細繩,放到一顆籃球裡面註,這究竟是如何折疊存放的?
這一切得從最簡單的 DNA 兩條序列開始,華生跟克里克在 1953 年提出 DNA 雙螺旋結構,也就是兩條序列有如雙螺旋般環繞,再往上一層,約 146 個 DNA 鹼基對環繞著組織蛋白 (histone) 構成 10 奈米大的核小體,有假說這如一串珠珠再纏繞形成 30 奈米的細胞質纖維(chromatin fibers),最後形成整個染色體,這中間的三級結構又是如何架構?
首先要想辦法搞清楚 DNA序列之間是否有結構上的特徵。但 DNA序列這麼多這麼長,該怎麼做起呢?
染色體上的結構特徵:拓撲結構域
2002 年美國哈佛大學 Job Dekker 等人發表染色體結構捕獲技術 (Chromosome Conformation Capture, 3C)1 ,主要利用甲醛將染色體空間中相近DNA序列固定住,然後透過限制酶剪開特定字串功能,將染色體裁切成小片段,進而把空間中相近DNA序列成對連接,最終透過 PCR 來觀察特定DNA片段在空間中是否相近交互。後續不斷有技術演進,其中 Hi-C 結合次世代定序技術 (next- generation sequencing)2與生物資訊分析方法,更易得到全基因組層面的成對 DNA 相近序列。
這些技術讓科學家進而推導出許多染色體結構上有趣的特徵,其中一個就是 TAD 拓撲結構域(Topology Associated Domain, 縮寫為TAD) 。

可將TAD想像成捲成一團的毛線球。圖/maxpixel
TAD 首先在 2012 年果蠅與人類的 Hi-C 論文中被提出來3,4,從 Hi-C 資料來看,TAD 區段內彼此交互的頻率會遠高於該區段外 (即空間上彼此會靠得比較相近,可以想像成一條線在這邊捲成一團,如毛線球),此性質可形成各自獨立的基因調控區域 ,隔開不該對應的啟動子 (promoter) 與增強子 (enhancer),避免錯誤的調控。
TAD可以依據不同生物特性,進一步區分成不同類別,如:活躍(active)、抑制(repressed)等。如何找出 TAD 的邊界、滿足上述性質,可以表達成一個數學問題,也就是給定一個 2 維數字矩陣 (Mi,j 為交互作用強度介於第 i 個與第 j 個基因體位置),如何找出區間集合 S,使得 (Mi,j > Mi,k | i,j ∈ Sx, k∈Sy)
究竟,TAD是染色體結構中真實存在的單位?還是 Hi-C 實驗中細胞群統計平均所得到的數學抽象概念?筆者本身參與了相關的研究5-7,剛好要試著回答這問題。
這一系列的研究,牽涉了相當多不同的技術。首先結合超高解析度光學顯微鏡 (dSTORM)與先進的 DNA標記技術 (Oligopaint),Cattoni 等人在單細胞層次觀察奈米尺度 TAD接觸行為,透過量化TAD內與外空間上接觸強弱機率,發現接觸強弱與細胞的型別、表觀的基因調控息息相關5。
接著 Szabo 等人進一步將顯微鏡光學標記(probe)標識在不同表觀類別的 TAD 上6,發現到,不同表觀 TAD 有著不同的行為:抑制 TAD 可觀察到為穩定的染色體組建單元,然而活躍 TAD 看不到空間中形成相對應的球聚架構 (圖一)。透過計算顯微鏡下奈米結構單元數量與當初所標記的抑制 TAD 數量一致,證明抑制 TAD 為染色體三級結構組成的基本奈米結構。另外,在顯微鏡底下可發現 TAD 內的兩點果真比TAD外的兩點距離來得短,再次證明 TAD 在三級上會形成一個較為緊密的架構,此外相同表觀類別的 TAD 在空間上較容易接觸。。
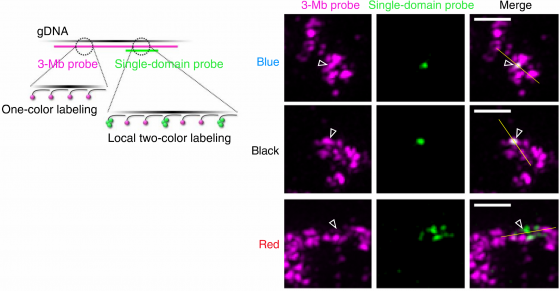
圖一. 左-粉紅色光學探針平均標示在 3M bps 的區段上,其中對於單一TAD另外標上綠色探針;右-依所標示的 TAD不同類別觀察 – 抑制 (Blue, Black) 和 活躍 (Red),可以看到單一TAD綠色探針在抑制TAD會形成一球,但在活躍TAD卻是分散的。圖/參考資料6 © The Authors, some rights reserved; exclusive licensee American Association for the Advancement of Science. Distributed under a Creative Commons Attribution NonCommercial License 4.0 (CC BY-NC) )
另外,染色體的結構在發育過程中也並不是固定一成不變的。Ogiyama 等人透過HiC實驗觀察染色體三級結構在果蠅胚胎發育的變化過程7,發現到染色體的結構是漸進式出現的:從前期無任何特定模式,到晚期形成 TAD 形式,這進展的過程與基因調控有密切的關係。
早期週期 1 到 8 染色體較為隨機的狀態,接著第一波 zygotic 轉錄時(週期 9~13),開始形成特定的活躍染色質環 (chromatin loop),接著才是TAD的形成,在更晚的週期 14,抑制的染色質環開始形成,最後跨整個染色體的長距離接觸在胚胎晚期系統地建立起來。
未來展望
近來利用超解析螢光顯微鏡8或電子顯微鏡9直接來觀察染色質結構也有所突破,打破先前教科書上理想的模型:「從10納米的串珠,往上架構成30納米的染色質纖維,再往上構成更大100~200納米的纖維」。科學家發現染色質會因細胞型別有各式不同的動態單元 (nucleosome clutches)8 或 5~20納米無序的鏈9。相信之後結合 Hi-C 與顯微鏡兩種資訊,人們終可以一解染色質結構功能之謎,並且回答更多的問題,例如:TAD 形成原因,其在演化上扮演的角色,與轉錄作用間的連結……等。
備註
- 若藍球直徑 24.5 公分, 則籃球直徑為細胞核的 40833 倍 (0.245/6 * 106 = 40833.33),就體積來說,一顆籃球約可以放入 408333 顆細胞核,把這些細胞核內染色體接起來 2*408333,若地球直徑為 40075 公里,共可繞 2*408333/(40075*1000)= 3397741 圈
參考資料
- Dekker, J., Rippe, K., Dekker, M. & Kleckner, N. Capturing Chromosome Conformation. Science 295, 1306–1311 (2002).
- Lieberman-Aiden, E. et al. Comprehensive mapping of long-range interactions reveals folding principles of the human genome. Science 326, 289–93 (2009).
- Sexton, T. et al. Three-dimensional folding and functional organization principles of the Drosophila genome. Cell 148, 458–72 (2012).
- Dixon, J. R. et al. Topological domains in mammalian genomes identified by analysis of chromatin interactions. Nature 485, 376–80 (2012).
- Valeri, A. et al. 2017. Single-cell absolute contact probability detection reveals chromosomes are organized by multiple low-frequency yet specific interactions. Nature Communications. 8, 1 (2017), 1753.
- Szabo, Q., Jost, D., Chang, J.-M., Cattoni, D., Papadopoulos, G., Bonev, B., Sexton, T., Gurgo, J., Jacquier, C., Nollmann, M., Bantignies, F. and Cavalli, G. 2018. TADs are 3D structural units of higher-order chromosome organization in Drosophila. Science Advances. 4, 2 (2018), eaar8082.
- Ogiyama, Y., Schuettengruber, B., Papadopoulos, G., Chang, J.-M. and Cavalli, G. 2018. Polycomb-Dependent Chromatin Looping Contributes to Gene Silencing during Drosophila Development. Molecular cell. 71, 1 (2018), 73–88.e5.
- Ricci, M. A., Manzo, C., García-Parajo, M. F. F., Lakadamyali, M. & Cosma, M. P. Chromatin fibers are formed by heterogeneous groups of nucleosomes in vivo. Cell 160, 1145–58 (2015).
- Ou, H. D. et al. ChromEMT: Visualizing 3D chromatin structure and compaction in interphase and mitotic cells. Science 357, eaag0025 (2017).
The post 長達兩公尺的遺傳資訊如何塞進六微米的細胞核?讓我們從染色體上的「拓樸結構域」談起 appeared first on PanSci 泛科學.